En la nota anterior construimos el cromosoma, que es la base para poder implementar el algoritmo genético. En esta nota agregaremos nuevo código a la clase cromosoma para establecer la forma en la que se evaluará una posible solución del problema y de esta manera se determine la función aptitud.
Nota: recordemos que la función aptitud es la forma de evaluar las soluciones representadas por los cromosomas. La que determina cuál de esas soluciones es la mejor.
Función aptitud
Recordemos que el problema del agente viajero consiste en realizar un recorrido circular de ciudades sin repetir ninguna. Sabemos que existen muchas formas de recorrer esas ciudades y que además existen algunos recorridos que cumplen con esas características, pero no es lo único que buscamos también queremos obtener la mejor solución posible.
Dado que la mejor solución al problema es la ruta más corta a través de nuestras ciudades, cada cromosoma representará un posible recorrido, y la función de aptitud indicará la distancia que necesita cubrir el agente viajero para pasar por todas las ciudades.
Distancia de una ciudad a otra
Para calcular la distancia entre dos puntos que se encuentran en cualquier lugar del sistema de coordenadas, la distancia queda determinada por:
Además debemos considerar que las soluciones pueden compartir subrecorridos, por lo que no es necesario calcularlos cada vez; así que es conveniente considerar en la implementación, una forma de guardar las distancias repetidas.
Nota: la implementación está escrita en Python.
Diccionario global para las distancias
#Diccionario global donde se guardan las distancias de una ciudad a otra.
distancias = {}
Métodos para agregar en la clase cromosoma
#Método que calcula la distancia de una ciudad a otra.
def distancia(self, cd1, cd2):
a,b = (cd1, cd2) if cd1.getid() < cd2.getid() else (cd2, cd1)
key = "%d-%d" % (a.getid(),b.getid())
if distancias.has_key(key): #El método has_key busca la distancia entre cd1 y cd2
dist= distancias[key]
else: """Si no encuentra la distancia entonces la calcula
y la guarda para futuras referencias"""
x= b.getx() - a.getx()
y= b.gety() - a.gety()
dist= sqrt(x**2 + y**2)
distancias[key]= dist
return dist
"""Método que calcula la aptitud asociada a un cromosoma, calcula la distancia
total de un recorrido de ciudades."""
def aptitud( self ):
"""Con el siguiente segmento de código se construye el recorrrido cirlular.
Ejemplo: para un recorrido de 3 ciudades queda de la siguiente forma [(1,2),(2,3),(3,1)]"""
apt = 0
orig= self.getciudades()
dest= list(orig)
dest.append(dest.pop())
for a,b in zip(orig,dest):
#El método zip genera una lista de tuplas a partir de dos listas dadas
apt += self.distancia(a,b)
return apt
#Método para comparar dos cromosomas.
def __cmp__(self, otro):
return int(self.aptitud() - otro.aptitud())
Función para ordenar las ciudades
#Método que ordena la población de mejor aptitud a peor aptitud.
def sort( self ):
self.__ciudades.sort()
Con estos métodos y funciones agregados a la implementación es posible determinar cuando un recorrido es mejor que otro, tomando como parámetro la menor distancia recorrida. Espero que hasta este punto esté siendo clara la explicación de la implementación, si tienes alguna duda puedes dejarla en los comentarios y con gusto la atenderé. ¡Hasta la siguiente entrega!

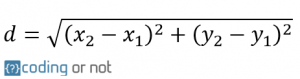
De nuevo mi agradecimiento por este trabajo. Hay alguna cosa que no entiendo bien.
Cuando pones
#Diccionario global donde se guardan las distancias de una ciudad a otra.
distancias = {}
¿En qué lugar hay que colocarlo? ¿Dentro de la clase cromosoma?
Igualmente, la función sort que sirve para ordenar, ¿Hay que ponerlo dentro de la clase cromosoma?
Gracias por tu respuesta
Hola Francisco
Gracias por tu comentario, me alegra saber que te ha sido útil.
El diccionario debes colocarlo en tu archivo principal, si es que tienes varios, pues es una variable global.
El método sort no pertenece a la clase cromosona, si tienes todo en un solo archivo puede ir fuera.
Disculpa la demora en la respuesta.
¡Saludos!